利用Python讀取fasta文件并進行一系列操作(上)
概述:
語言:python3.8
模塊:pysam,collections
可選:jupyter noebook
整體思路:將fasta格式的基因原始數據處理為方便讀寫的txt格式并進行操作
步驟:
1. 獲取自己的fasta文件(這里我將從NCBI上下載人類的ABO基因參考序列的fasta文件為例)
?
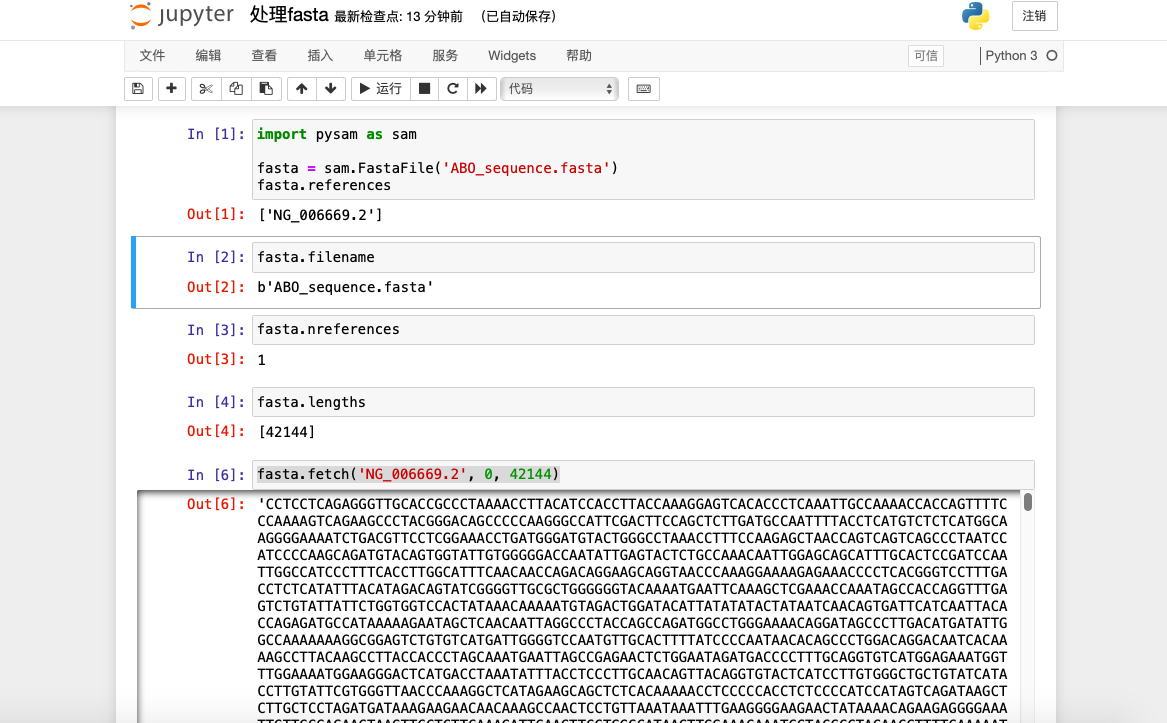
2. 利用pysam模塊的FastaFile函數讀取fasta,之后即可獲取fasta的基本信息:filename 文件名,references 染色體編號(因為這里我下載的是ABO基因的fasta,所以不是染色體編號),nreferences 染色體數,lengths 每條染色體長度(這個非常重要!)
?
3. 通過判斷輸出的長度,我們知道了這個ABO基因的長度值為42144,我們再利用fetch函數獲取ABO基因堿基序列(注意索引是從0開始)
?
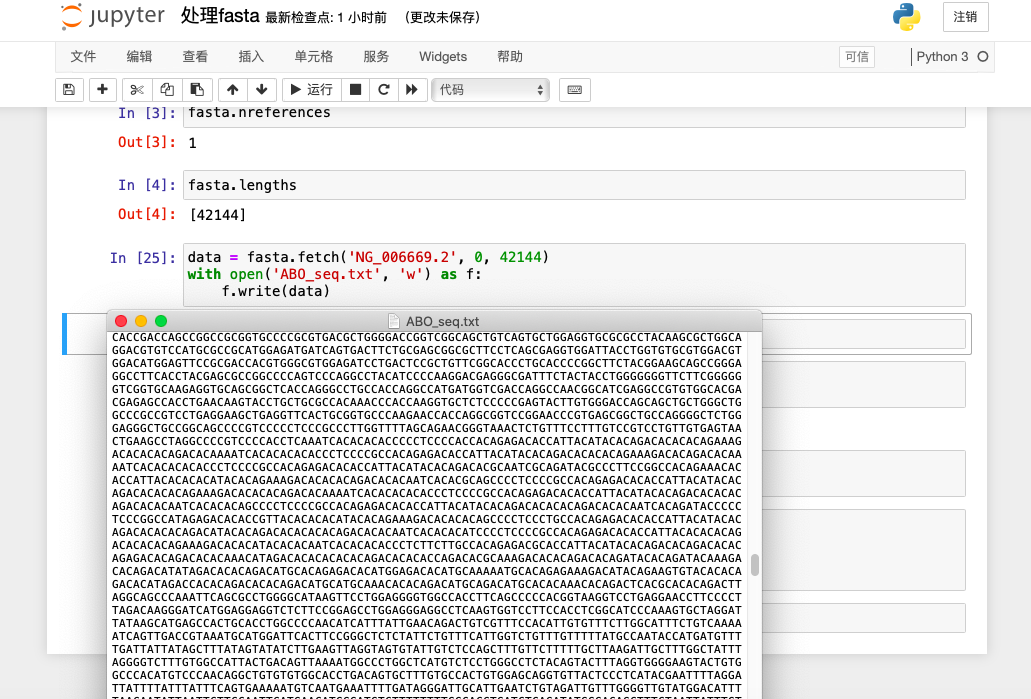
4. 我們的最終目的是要輸出txt,所以就需要將我們的堿基序列寫到txt上
?
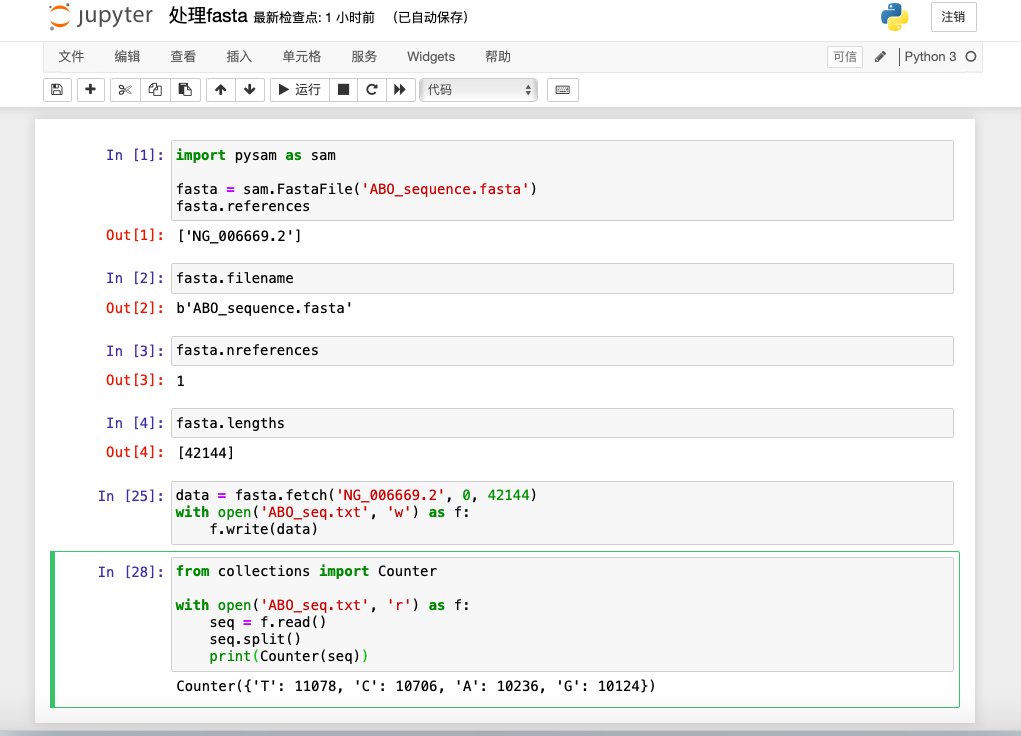
5. 我們可以利用我們輸出的txt來做點事情,例如看看這個序列中各種堿基數量,先讀取文件,再處理為列表,利用collection模塊的Counter函數來輸出結果
?
更多操作將在下篇解鎖!
語言:python3.8
模塊:pysam,collections
可選:jupyter noebook
整體思路:將fasta格式的基因原始數據處理為方便讀寫的txt格式并進行操作
步驟:
1. 獲取自己的fasta文件(這里我將從NCBI上下載人類的ABO基因參考序列的fasta文件為例)
?
2. 利用pysam模塊的FastaFile函數讀取fasta,之后即可獲取fasta的基本信息:filename 文件名,references 染色體編號(因為這里我下載的是ABO基因的fasta,所以不是染色體編號),nreferences 染色體數,lengths 每條染色體長度(這個非常重要!)
?
3. 通過判斷輸出的長度,我們知道了這個ABO基因的長度值為42144,我們再利用fetch函數獲取ABO基因堿基序列(注意索引是從0開始)
?
4. 我們的最終目的是要輸出txt,所以就需要將我們的堿基序列寫到txt上
?
5. 我們可以利用我們輸出的txt來做點事情,例如看看這個序列中各種堿基數量,先讀取文件,再處理為列表,利用collection模塊的Counter函數來輸出結果
?
更多操作將在下篇解鎖!


1 個回復
贊同來自:
要回復問題請先登錄或注冊