利用Python讀取fasta文件并進行一系列操作(二)
概述:
本節目標:通過上一節所輸出的txt輸出ABO蛋白以及ABO基因的外顯子fasta文件
語言:python3.8
模塊:Biopython, ssl
可選:jupyter
整體思路:通過ncbi獲取ABO基因外顯子位置(爬蟲),并讀取txt,根據位置信息獲取外顯子序列,再通過外顯子
序列輸出mRNA序列以及蛋白序列
前排提示:本教程不管生物,有知識盲區自己補
步驟:
1.?設置ssl,要不debug后總會有bug
2. 從這步開始調用Biopython,設置郵箱(不要瞎填!!瞎填還不如不填!!!)
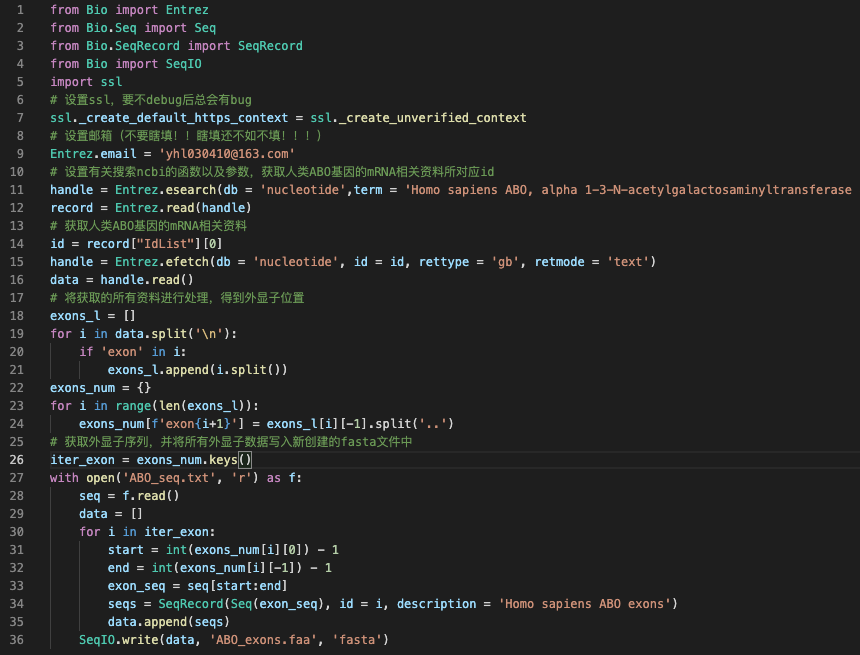
3.?設置有關搜索ncbi的函數以及參數,獲取人類ABO基因的mRNA相關資料所對應id
4.?獲取人類ABO基因的mRNA相關資料
5.?將獲取的所有資料進行處理,得到外顯子位置
6.?獲取外顯子序列,并將所有外顯子數據寫入新創建的fasta文件中
代碼:
?
結果展示:
?
獲取蛋白序列繼續往下看:
接上面步驟:
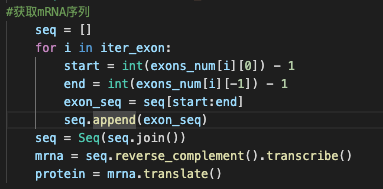
7. 將獲取的外顯子序列用Seq函數轉為biopython類型
8. 將外顯子倒序并轉錄
9. 將轉錄出的mRNA序列翻譯為蛋白質序列
代碼:
?
?
本節目標:通過上一節所輸出的txt輸出ABO蛋白以及ABO基因的外顯子fasta文件
語言:python3.8
模塊:Biopython, ssl
可選:jupyter
整體思路:通過ncbi獲取ABO基因外顯子位置(爬蟲),并讀取txt,根據位置信息獲取外顯子序列,再通過外顯子
序列輸出mRNA序列以及蛋白序列
前排提示:本教程不管生物,有知識盲區自己補
步驟:
1.?設置ssl,要不debug后總會有bug
2. 從這步開始調用Biopython,設置郵箱(不要瞎填!!瞎填還不如不填!!!)
3.?設置有關搜索ncbi的函數以及參數,獲取人類ABO基因的mRNA相關資料所對應id
4.?獲取人類ABO基因的mRNA相關資料
5.?將獲取的所有資料進行處理,得到外顯子位置
6.?獲取外顯子序列,并將所有外顯子數據寫入新創建的fasta文件中
代碼:
?
結果展示:
?
獲取蛋白序列繼續往下看:
接上面步驟:
7. 將獲取的外顯子序列用Seq函數轉為biopython類型
8. 將外顯子倒序并轉錄
9. 將轉錄出的mRNA序列翻譯為蛋白質序列
代碼:
?
?



1 個回復
贊同來自:
要回復問題請先登錄或注冊