利用WeGene WGS給出的VCF文件輸出類似WeGene芯片數據txt
概述:
編程語言:Python3.8
模塊:pyvcf csv
可選:jupyter
整體思路:識別WeGene芯片數據txt的文件特征,讀取vcf文件并根據其中內容獲取所需數據并寫入到txt中
前排提示:強烈建議買一個讀寫速度快一點而且至少是128GB或以上的U盤,當然我是直接買了個1T的移動硬盤
?
步驟:
1. 通過觀察微基因芯片測試txt結果,我們可以得知重要信息分別為:RSID chromosome position genotype,并且我們可以發現每列之間的分隔符是一個tab縮進,所以接下來的問題就轉化成如何獲取這四種信息并且以分隔符為tab縮進形式輸出
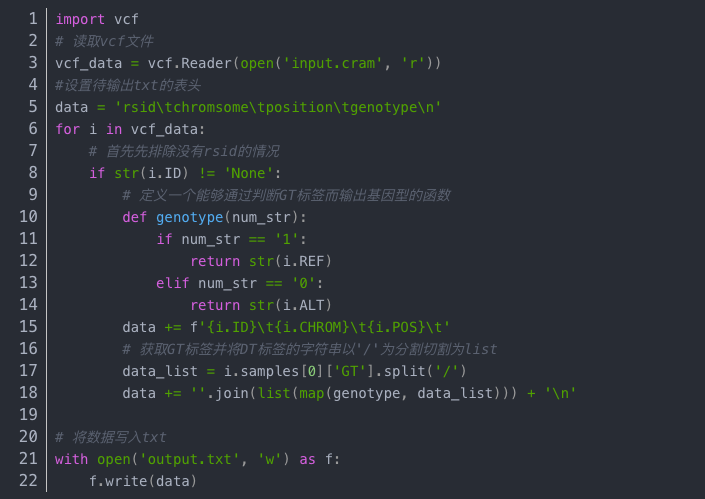
2. 眾所周知vcf文件里的數據包括ID CHROM POS 參考序列和突變方向,而vcf中又有GT標簽來表示是雜合還是純合,是突變還是未突變。所以得出結論:我們可以直接調用pyvcf模塊,并讀取vcf,遍歷每行的內容,同時直接用ID POS CHROM標簽來獲取所需的RSID chromosome position三種數據,再通過讀取GT標簽,原始型REF和突變型ALT來確定這個位點的genotype。注意:這幾種標簽的數據類型如下表
標簽 |?類型
ID |?str
CHROM | int
POS | int
ID | str
GT | str
REF | str
ALT | list
GT標簽說明:示例結構'0/1',其中0表示原始型,1表示突變
代碼:
?
結果展示:
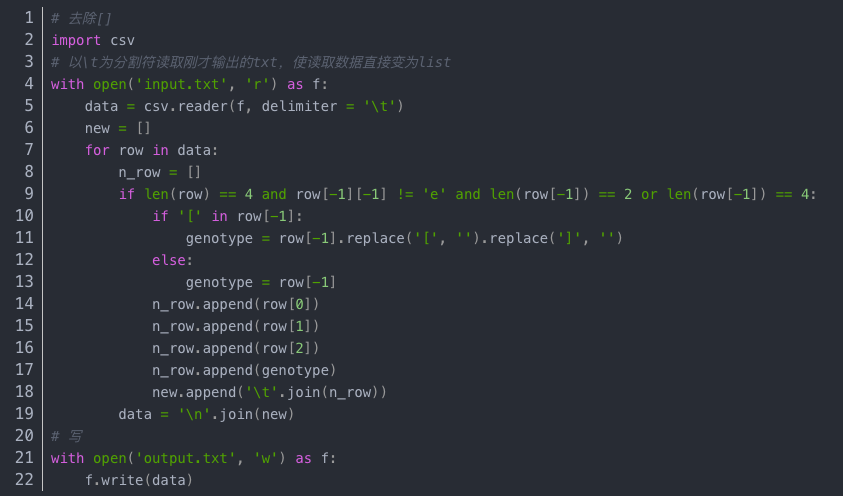
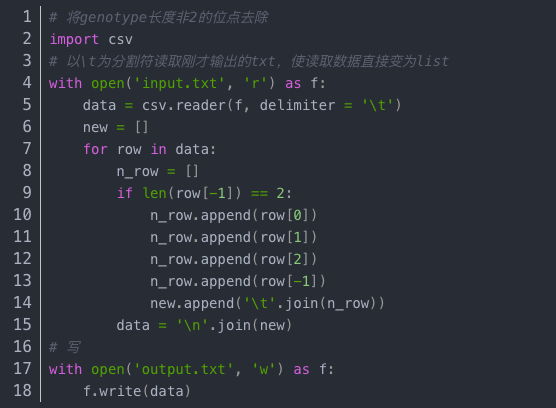
3. 從我們輸出的結果可以看出還有些問題,由于ALT標簽輸出的是list所以文件中會存在[]字樣,并且由于讀取的位點中并不是所有位點的genotype長度都是2,而微基因的數據中的genotype長度都是2,所以去除[]的同時還要去除那些genotype長度非2的位點
代碼:
?
?
編程語言:Python3.8
模塊:pyvcf csv
可選:jupyter
整體思路:識別WeGene芯片數據txt的文件特征,讀取vcf文件并根據其中內容獲取所需數據并寫入到txt中
前排提示:強烈建議買一個讀寫速度快一點而且至少是128GB或以上的U盤,當然我是直接買了個1T的移動硬盤
?
步驟:
1. 通過觀察微基因芯片測試txt結果,我們可以得知重要信息分別為:RSID chromosome position genotype,并且我們可以發現每列之間的分隔符是一個tab縮進,所以接下來的問題就轉化成如何獲取這四種信息并且以分隔符為tab縮進形式輸出
2. 眾所周知vcf文件里的數據包括ID CHROM POS 參考序列和突變方向,而vcf中又有GT標簽來表示是雜合還是純合,是突變還是未突變。所以得出結論:我們可以直接調用pyvcf模塊,并讀取vcf,遍歷每行的內容,同時直接用ID POS CHROM標簽來獲取所需的RSID chromosome position三種數據,再通過讀取GT標簽,原始型REF和突變型ALT來確定這個位點的genotype。注意:這幾種標簽的數據類型如下表
標簽 |?類型
ID |?str
CHROM | int
POS | int
ID | str
GT | str
REF | str
ALT | list
GT標簽說明:示例結構'0/1',其中0表示原始型,1表示突變
代碼:
?
結果展示:
3. 從我們輸出的結果可以看出還有些問題,由于ALT標簽輸出的是list所以文件中會存在[]字樣,并且由于讀取的位點中并不是所有位點的genotype長度都是2,而微基因的數據中的genotype長度都是2,所以去除[]的同時還要去除那些genotype長度非2的位點
代碼:
?
?



4 個回復
如果存在插入缺失位點,那么需要在sed+sort之前用其他方法將其轉化為“I”或“D”。
贊同來自:
贊同來自:
贊同來自: [已注銷]
贊同來自:
要回復問題請先登錄或注冊