利用AlphaFold進行蛋白質結構預測
AlphaFold是DeepMind公司下的一個預測蛋白質結構的AI,這個公司下的AI還有我們所熟悉的AlphaGo。它對大部分蛋白質結構的預測與真實結構只差一個原子的寬度,達到了人類利用冷凍電子顯微鏡等復雜儀器觀察預測的水平。該程序利用Python寫成。今天我們將利用它的云端Jupyter Notebook根據人的紅細胞H抗原的氨基酸序列對人的紅細胞H抗原的蛋白結構進行預測。
?
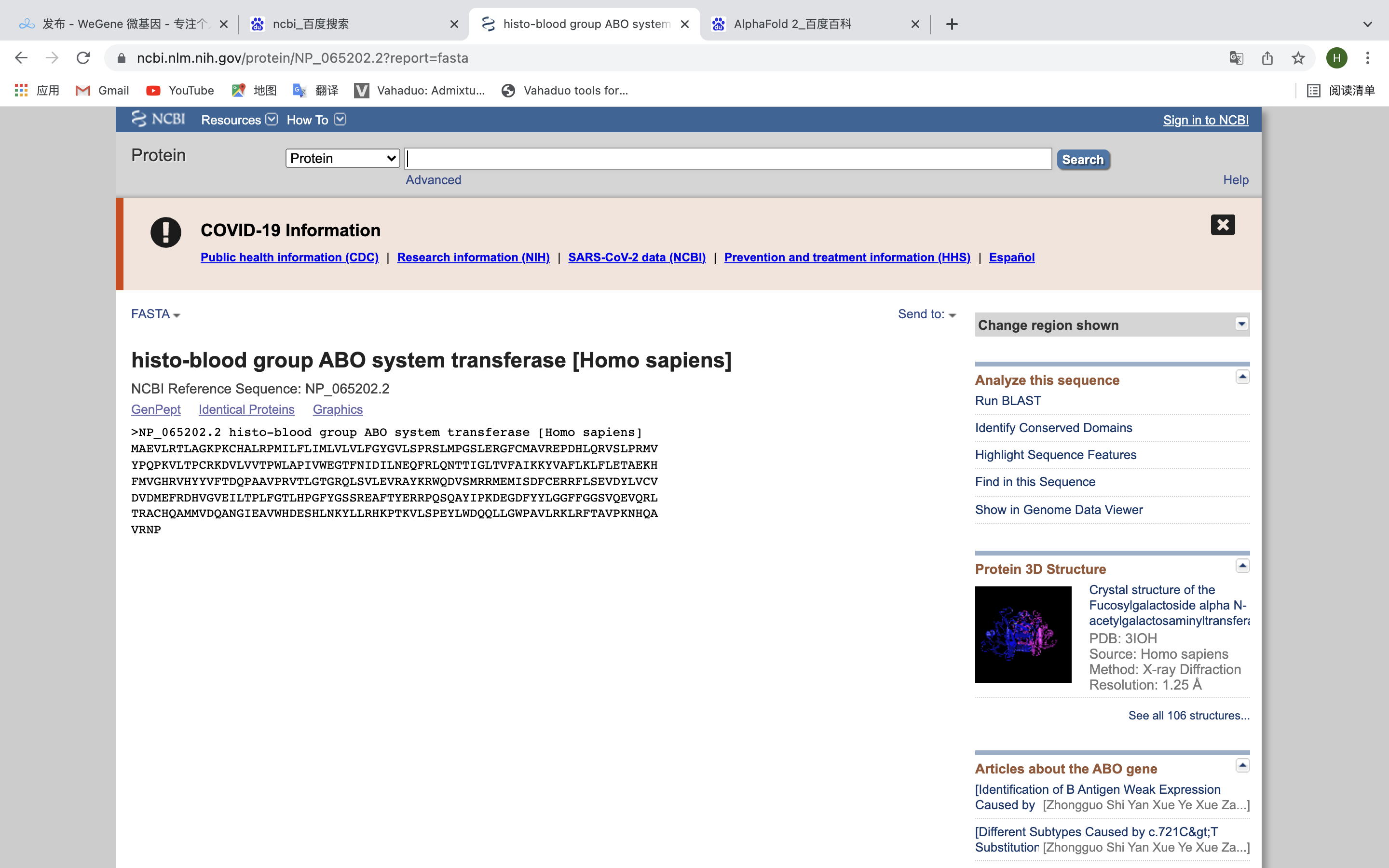
首先先打開NCBI找到相關氨基酸序列,把它copy下來
?
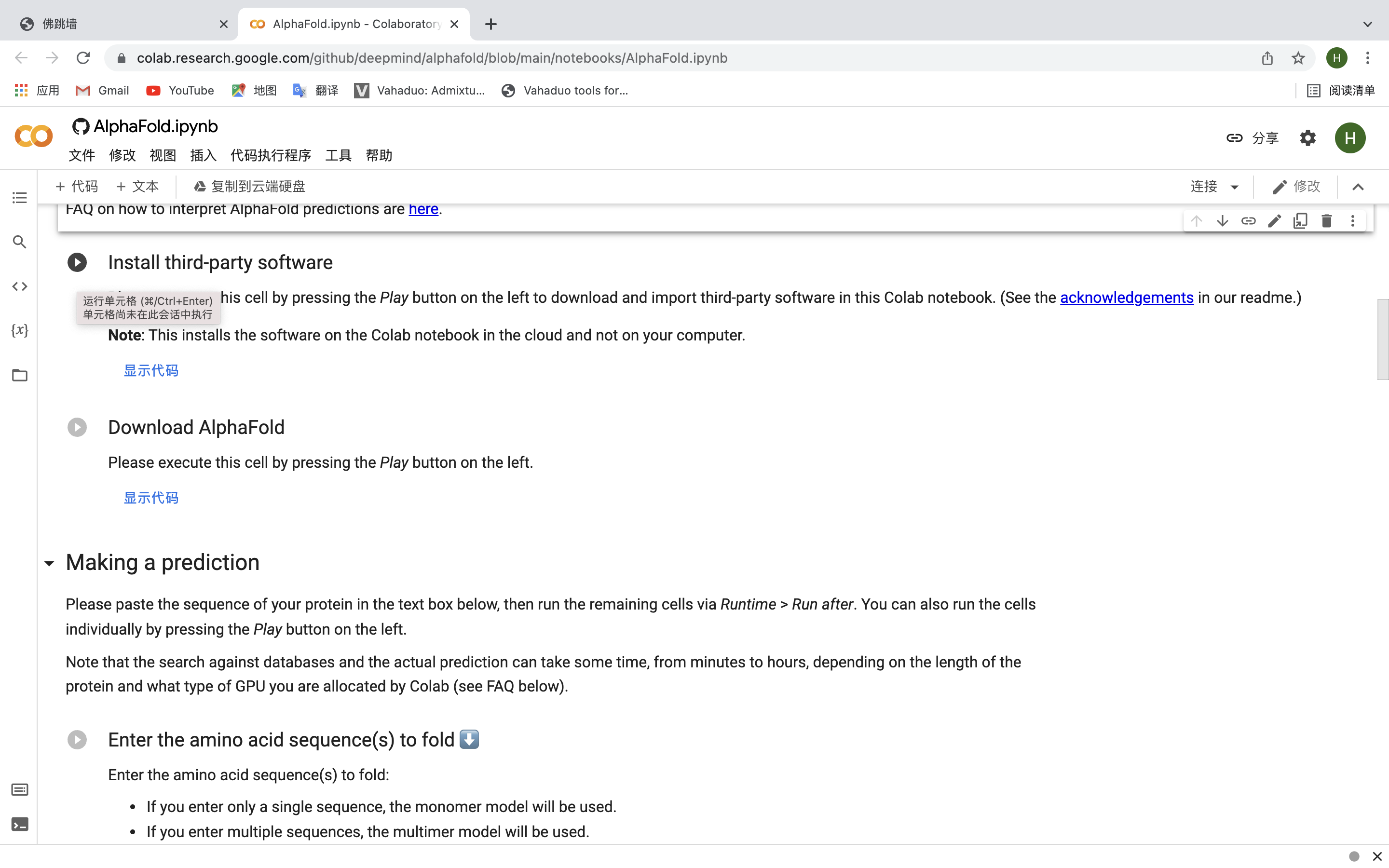
之后打開這個網址:?https://colab.research.google.com/github/deepmind/alphafold/blob/main/notebooks/AlphaFold.ipynb
?
之后首先按順序運行前兩單元,安裝運行環境(這個安裝過程只會安裝到云端,不會安裝到本地,放心)
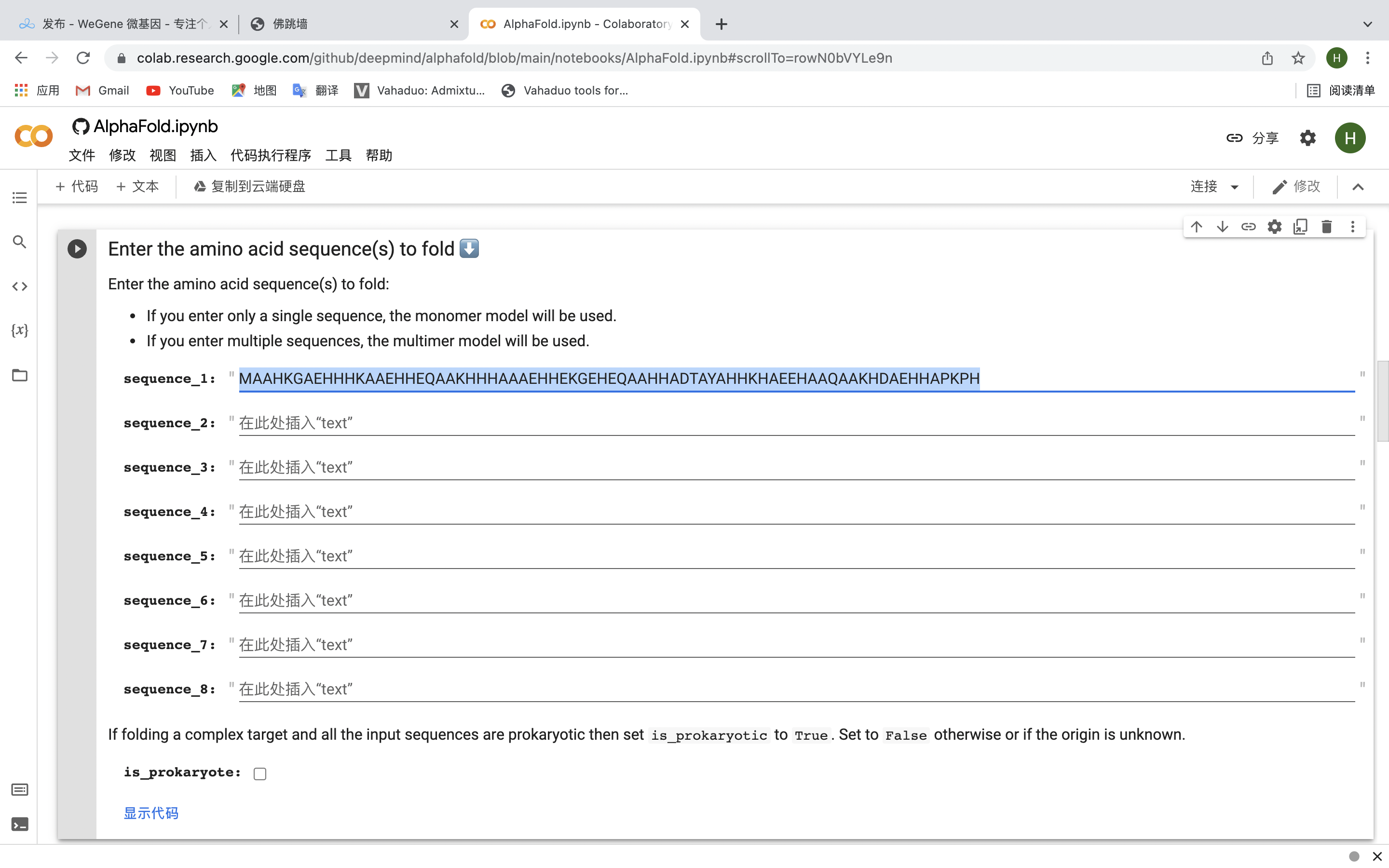
再將剛才copy的氨基酸序列放在圖中選中的部分,點運行(注意如果預測氨基酸序列來自于原核生物記得勾選is_prokaryote)
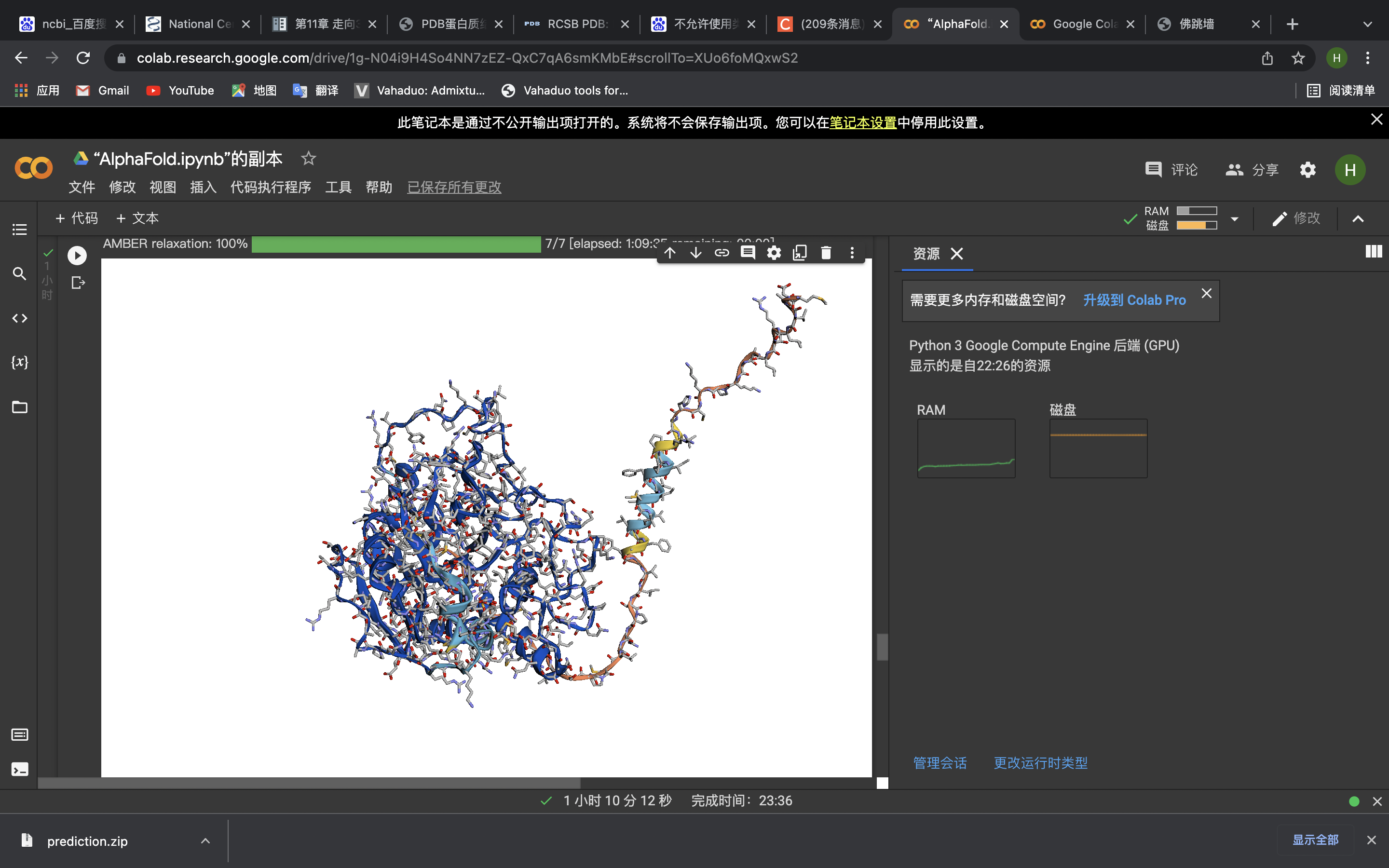
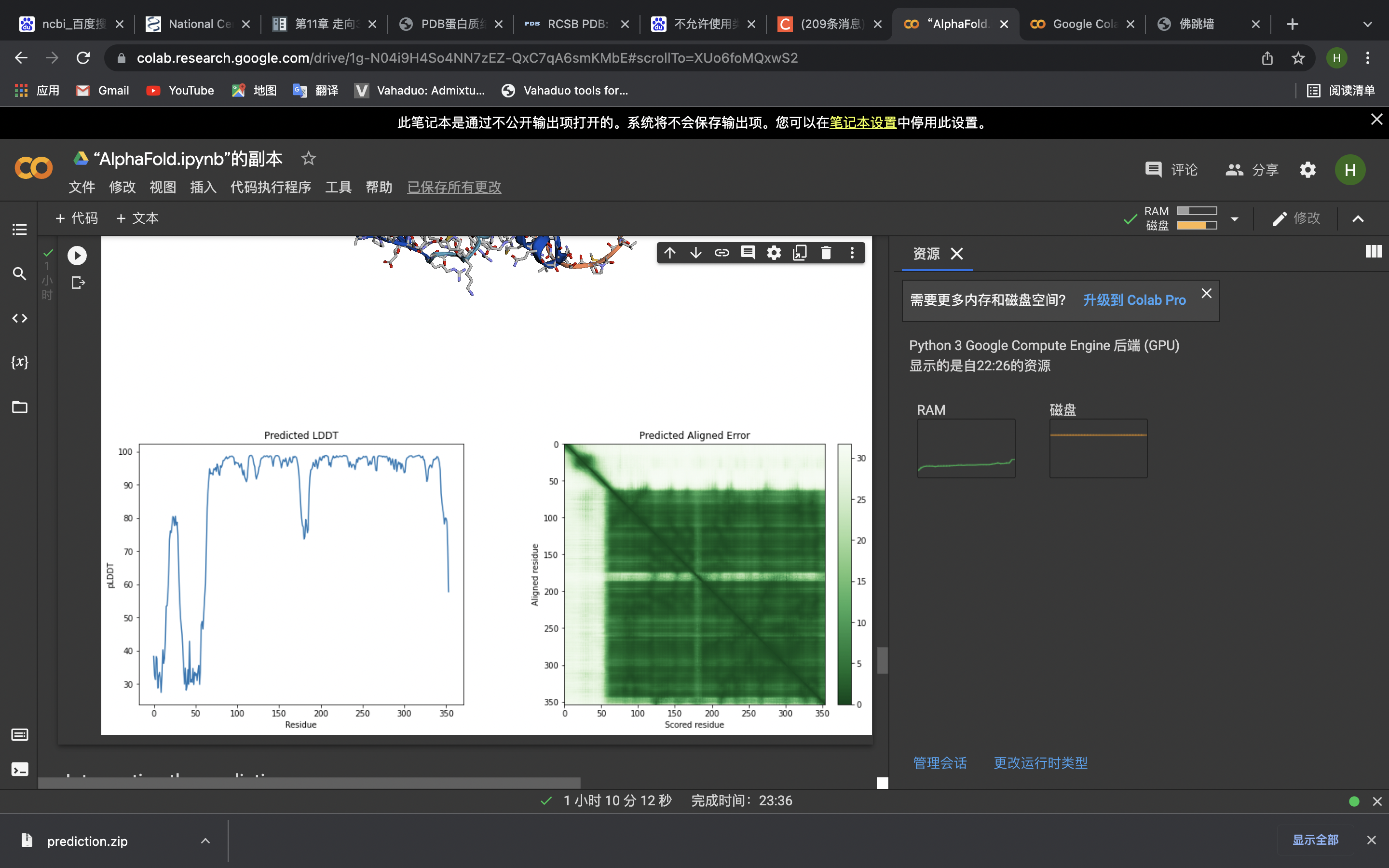
之后接著向下運行完所有單元(時間會比較長,因為會和數據庫對比)
運行完會自動下載預測好的蛋白質結構的PDB文件
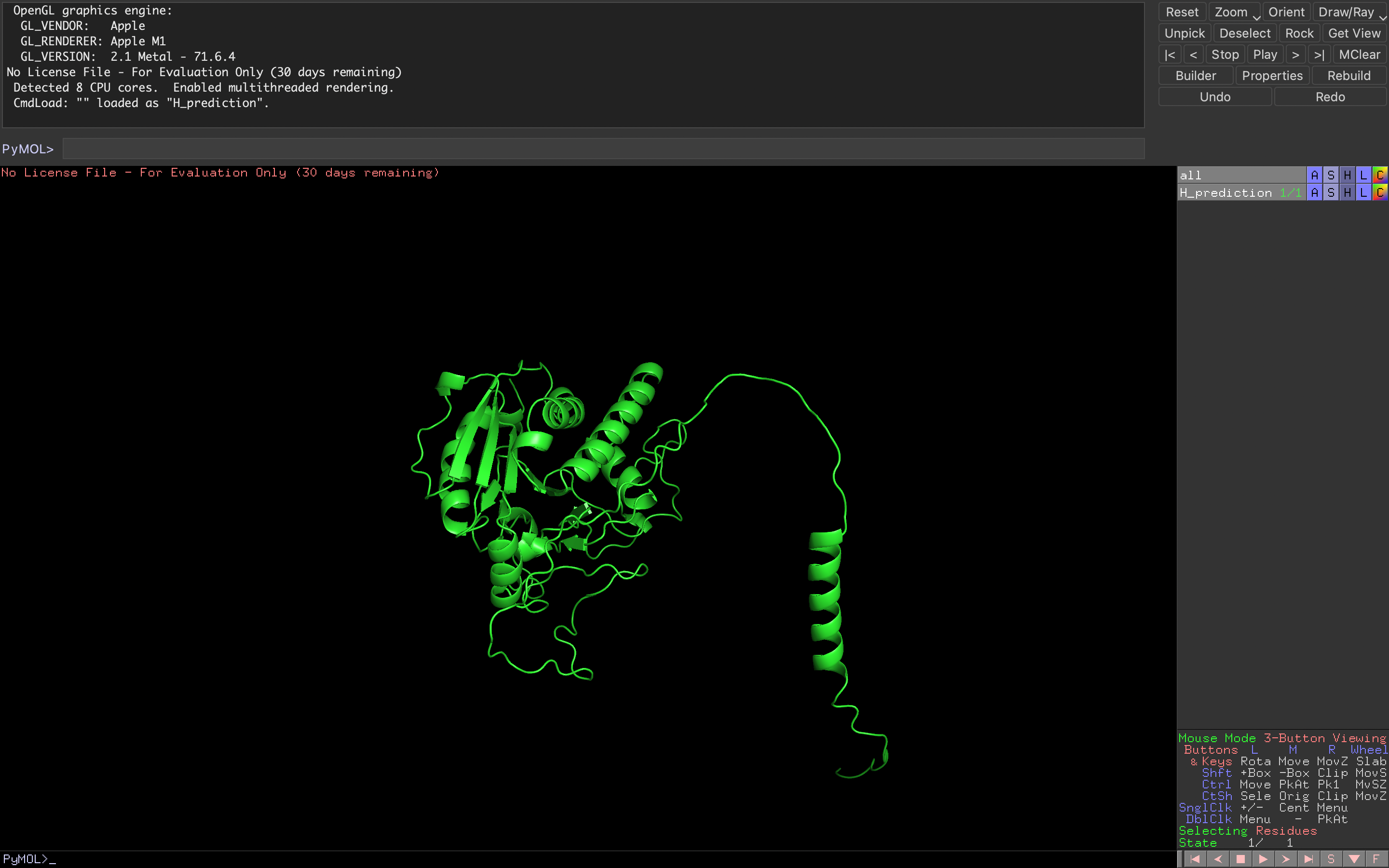
之后可以利用pyMOL軟件在線下打開PDB文件,輸出圖片
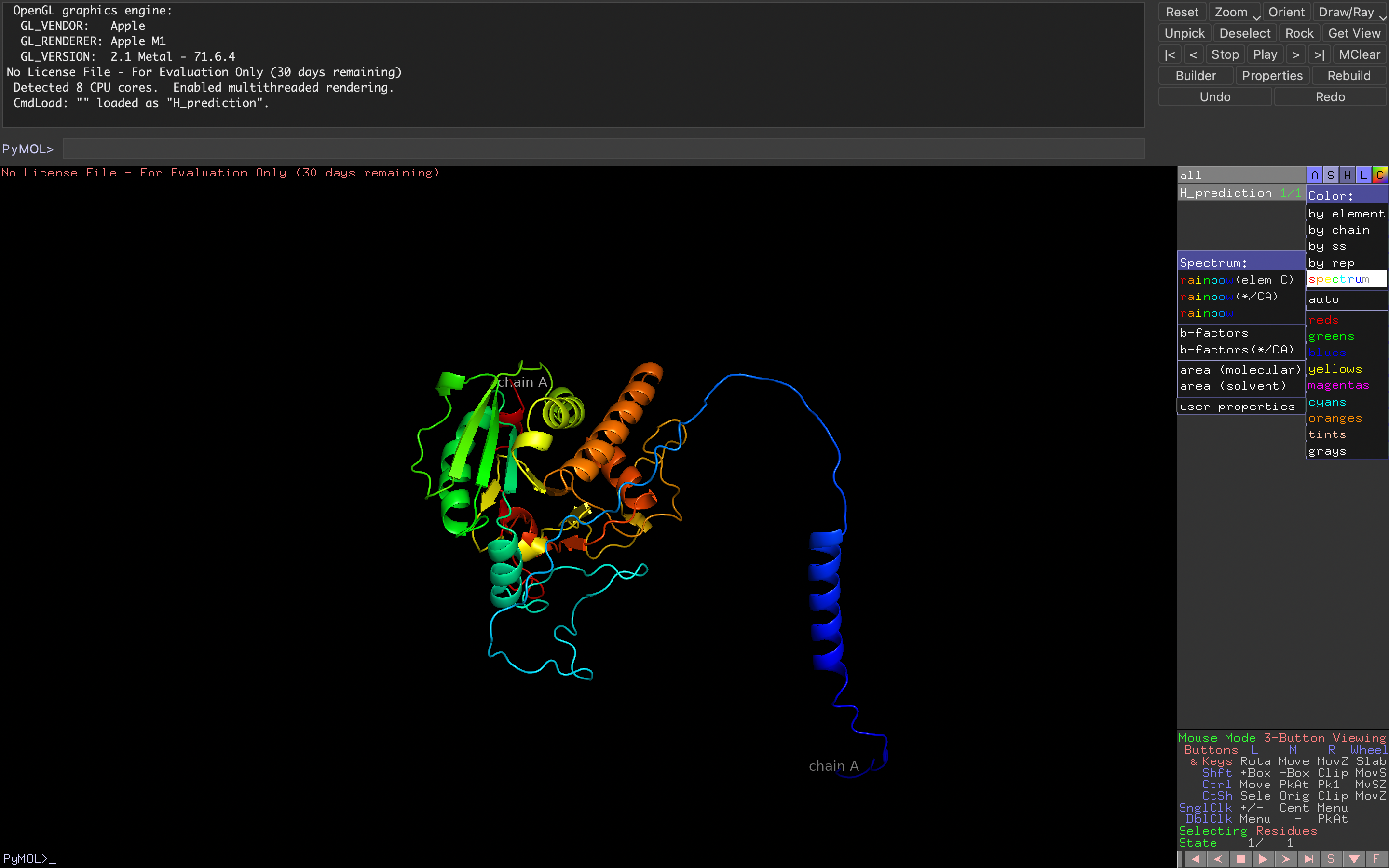
你還可以通過這個軟件改變顏色
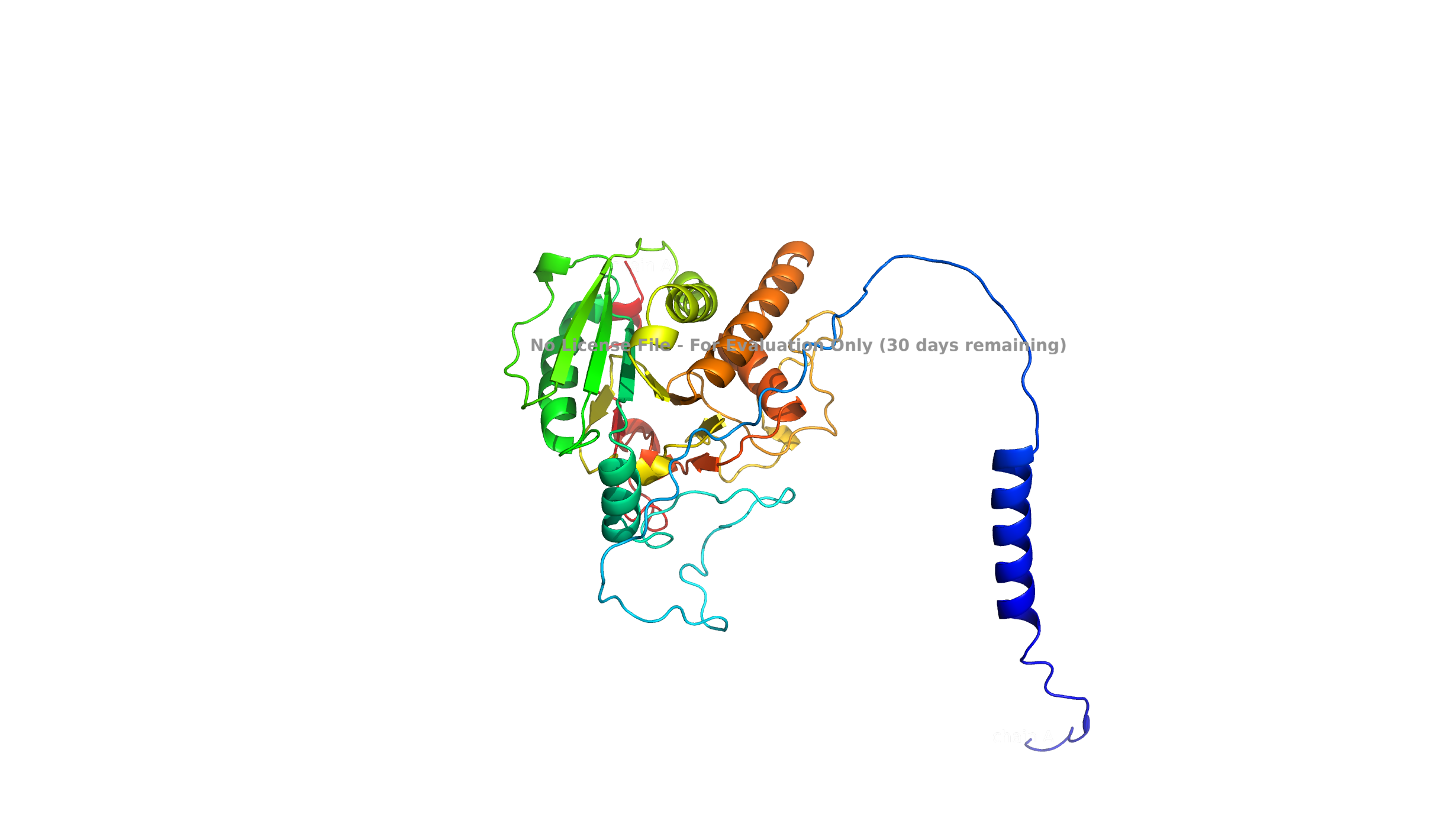
結果展示:
?
現在我正在研究如何把PDB文件轉換為STL文件,之后或許能將預測結構用3D打印機打印出來,敬請期待~
?
?
首先先打開NCBI找到相關氨基酸序列,把它copy下來
?
之后打開這個網址:?https://colab.research.google.com/github/deepmind/alphafold/blob/main/notebooks/AlphaFold.ipynb
?
之后首先按順序運行前兩單元,安裝運行環境(這個安裝過程只會安裝到云端,不會安裝到本地,放心)
再將剛才copy的氨基酸序列放在圖中選中的部分,點運行(注意如果預測氨基酸序列來自于原核生物記得勾選is_prokaryote)
之后接著向下運行完所有單元(時間會比較長,因為會和數據庫對比)
運行完會自動下載預測好的蛋白質結構的PDB文件
之后可以利用pyMOL軟件在線下打開PDB文件,輸出圖片
你還可以通過這個軟件改變顏色
結果展示:
?
現在我正在研究如何把PDB文件轉換為STL文件,之后或許能將預測結構用3D打印機打印出來,敬請期待~
?



4 個回復
贊同來自: 蝕月
贊同來自:
贊同來自:
贊同來自:
要回復問題請先登錄或注冊