關于基于PRS的專業版微解讀代碼和說明
多基因風險評分分析(Polygenic Risk Score)是一個用來評估個體患某種疾病風險的方法,它是通過綜合GWAS統計數據的基因型效應值來計算的。
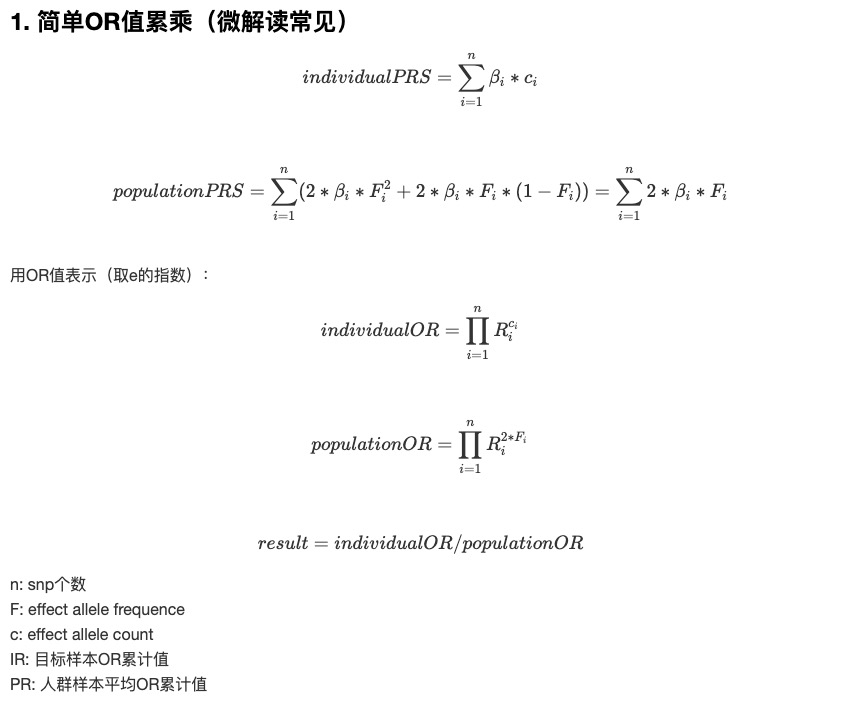
計算公式如下圖所示:
?
做微解讀時候應該注意:
1. 我們最終要給出個體風險值相對人群平均風險值的倍數關系,所以最后結果一定是累積OR值相除,而不是累積beta值相除,不然結果會產生負數。
2. 收集GWAS文獻中的snp信息時,注意文章給出的effect allele是否是基因組正鏈的,如果不是需要用堿基互補規則修正過來。
3. 每個位點都需要處理no call或不存在的情況,一般都是用maf值來折算,位點的人群maf可以從公共數據庫dbsnp中查得,一般選東亞人群的。
?
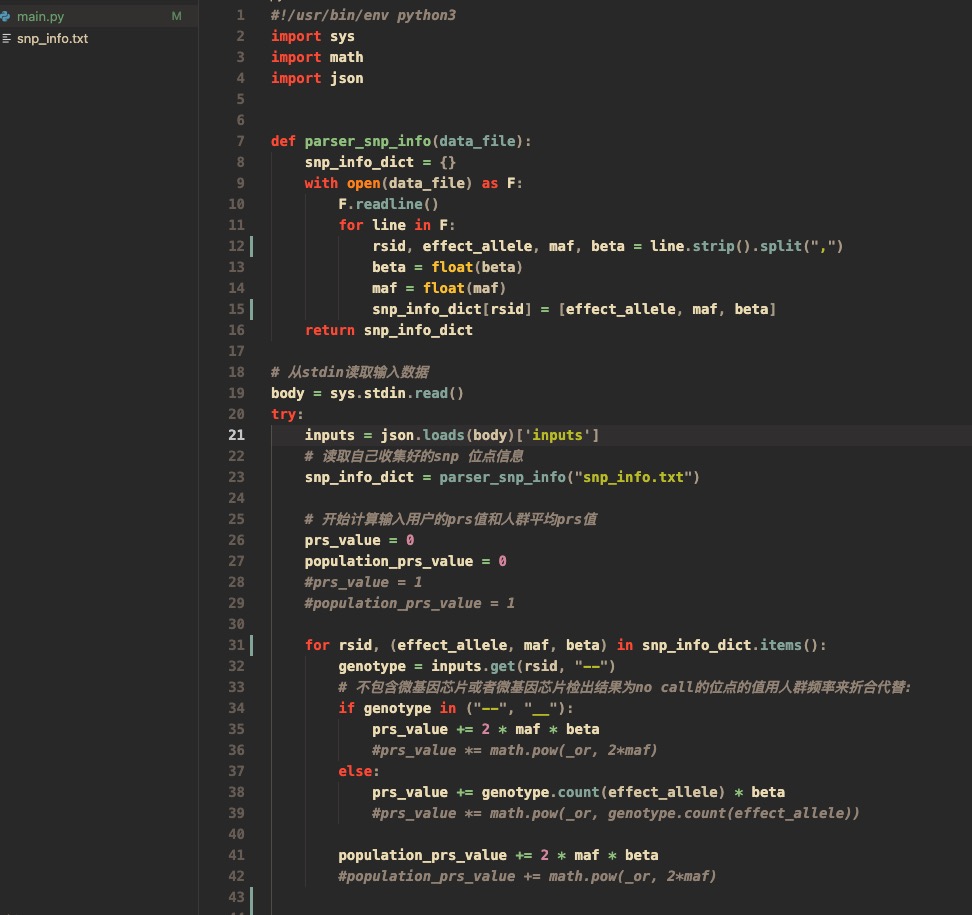
這里給一個示例代碼給大家參考下:
?
代碼說明:
1. 正常情況下,下載該源碼,只需將snp_info.txt中的snp信息修改成自己收集的信息(格式和列順序不能改),不做其他更改即可使用。
2. 代碼給出的是用beta值來計算,如果GWAS文章給的是OR值, 可先將其轉化為beta值(beta = ln(OR)),也可以直接使用OR值計算(示例代碼里也有,只是被注釋掉了)
3. 另外雖然微解讀支持python調用第三方庫,但建議只使用python標準庫來解決簡單的數據處理問題,這樣有利于微解讀代碼審核者發現問題。
?
代碼下載地址:https://github.com/TuBieJun/wegene_weijiedu_example
?
希望大家都能做出自己的專業版微解讀!
?
計算公式如下圖所示:
?
做微解讀時候應該注意:
1. 我們最終要給出個體風險值相對人群平均風險值的倍數關系,所以最后結果一定是累積OR值相除,而不是累積beta值相除,不然結果會產生負數。
2. 收集GWAS文獻中的snp信息時,注意文章給出的effect allele是否是基因組正鏈的,如果不是需要用堿基互補規則修正過來。
3. 每個位點都需要處理no call或不存在的情況,一般都是用maf值來折算,位點的人群maf可以從公共數據庫dbsnp中查得,一般選東亞人群的。
?
這里給一個示例代碼給大家參考下:
?
代碼說明:
1. 正常情況下,下載該源碼,只需將snp_info.txt中的snp信息修改成自己收集的信息(格式和列順序不能改),不做其他更改即可使用。
2. 代碼給出的是用beta值來計算,如果GWAS文章給的是OR值, 可先將其轉化為beta值(beta = ln(OR)),也可以直接使用OR值計算(示例代碼里也有,只是被注釋掉了)
3. 另外雖然微解讀支持python調用第三方庫,但建議只使用python標準庫來解決簡單的數據處理問題,這樣有利于微解讀代碼審核者發現問題。
?
代碼下載地址:https://github.com/TuBieJun/wegene_weijiedu_example
?
希望大家都能做出自己的專業版微解讀!
?



3 個回復
贊同來自:
贊同來自:
贊同來自:
要回復問題請先登錄或注冊