HGG.SNV數據庫批量獲取信息
HGG.SNV是一個了解不同人群中人類單核苷酸變異的進化和醫學意義的數據庫。這個數據庫的亮點是它綜合了很多項目的各種測序數據,其中包括HGDP、HapMap、gnomAD.genomes、SGDP等項目數據。其中包括220147份基因組數據,古人基因組1018份,覆蓋了977個族群的268890619個位點。另一個亮點是有多種線上工具,操作方便。
?
網站鏈接:https://www.pggsnv.org/
?
但是當我們要查詢的數據量十分龐大時會比較費時間,所以我們寫一段python腳本來批量獲取數據。
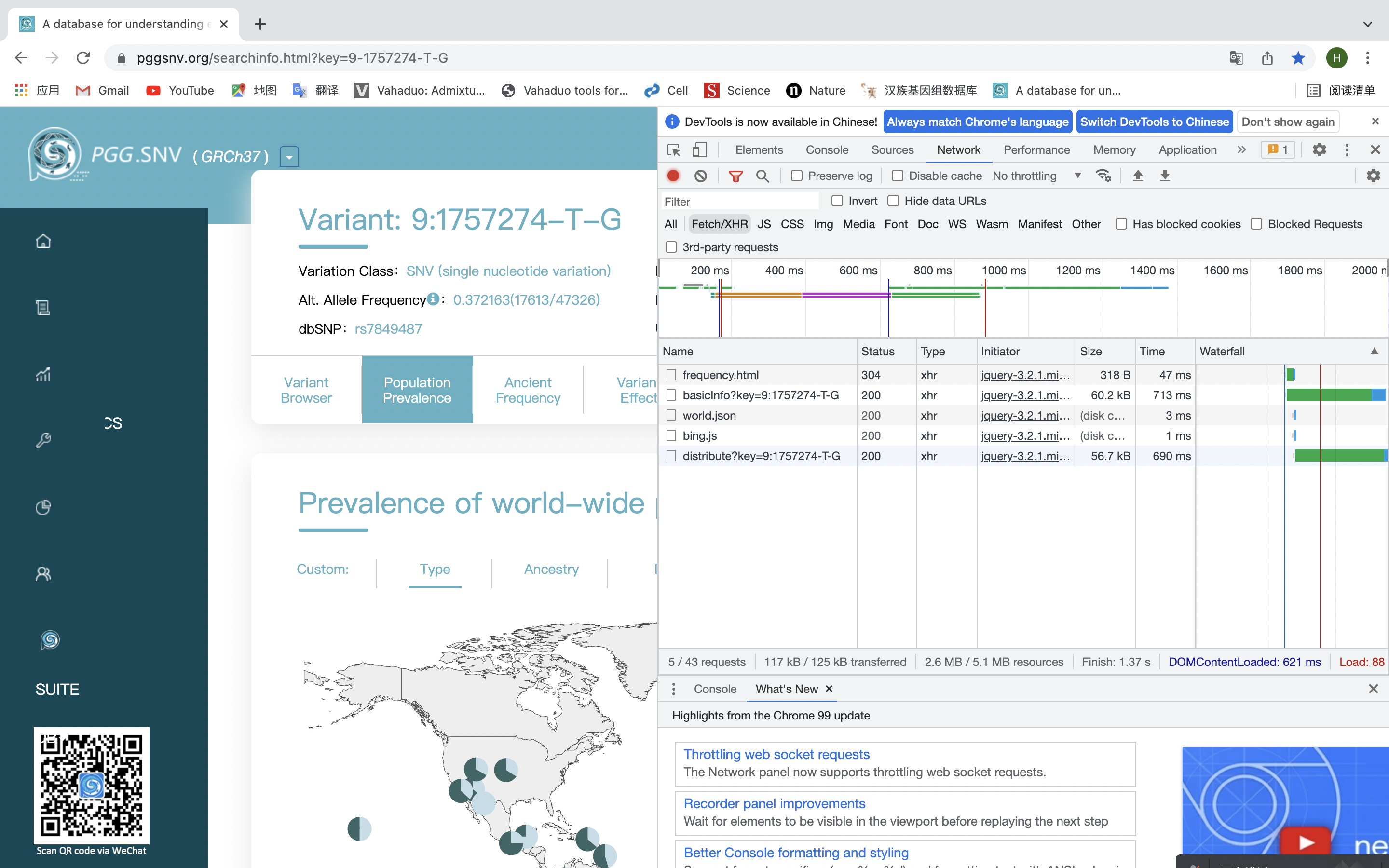
首先使用chrome對該網站的基本情況進行分析。使用Chrome的開發者工具對傳輸數據進行監控,并將傳輸數據篩選為fetch和XHR模式,發現該網站的前后端是通過json傳輸的,屬于前后端分離的情況,所以我們可以直接向網站拉去請求獲取json。
?
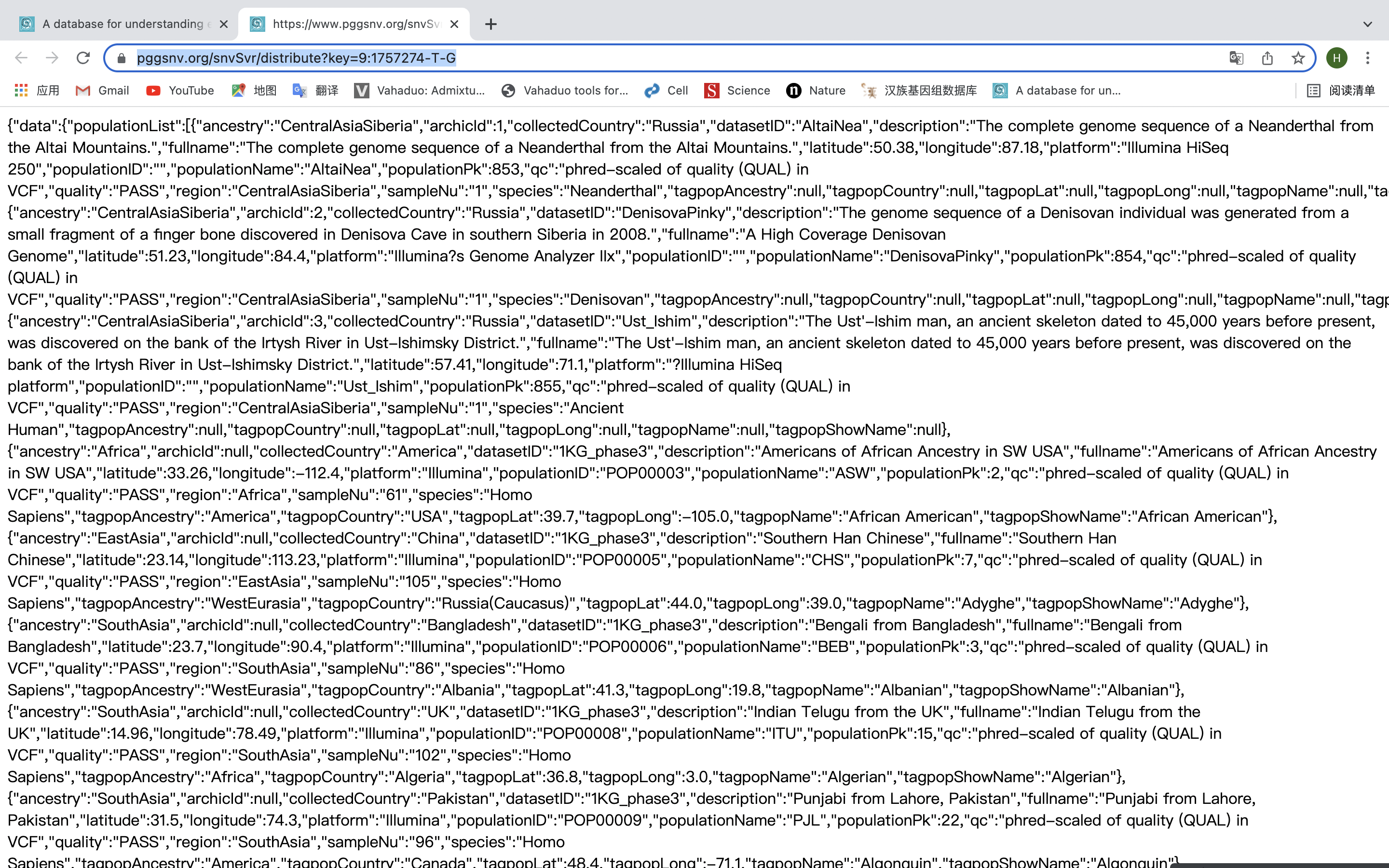
我們將其中的json打開,發現里面確實含有位點相關信息,所以雙擊該json文件獲取url。
?
url:https://www.pggsnv.org/snvSvr/distribute?key=1:231557623-G-C
?
接著我們對url進行解析。通過對比已知位點數據可知該url中的key應該是以這樣的格式組成的:染色體號:堿基對位置-ref-alt
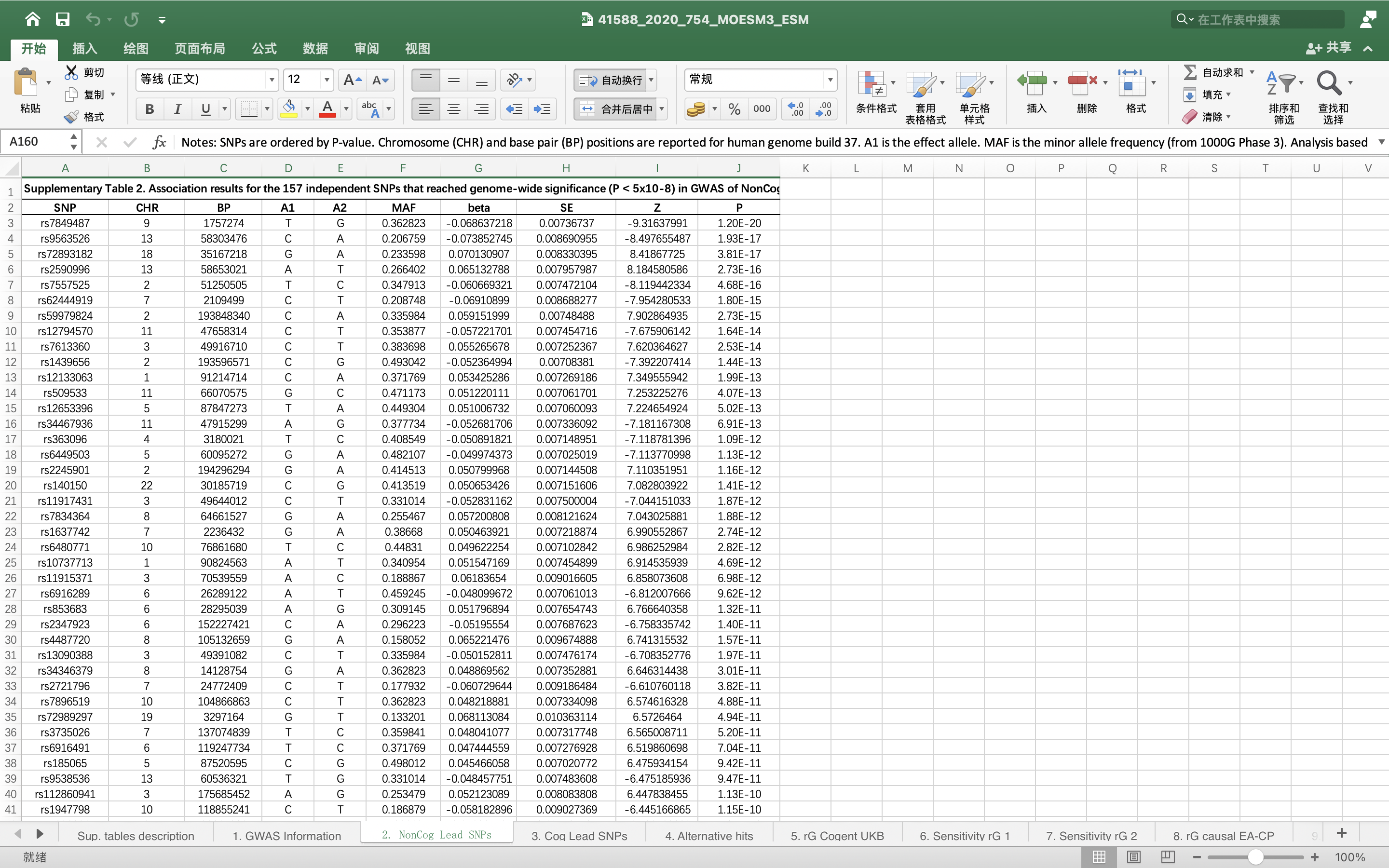
我們以《Nature-Genetics》期刊上的《Investigating the genetic architecture of noncognitive skills using GWAS-by-subtraction》【1】這篇文章的數據為例
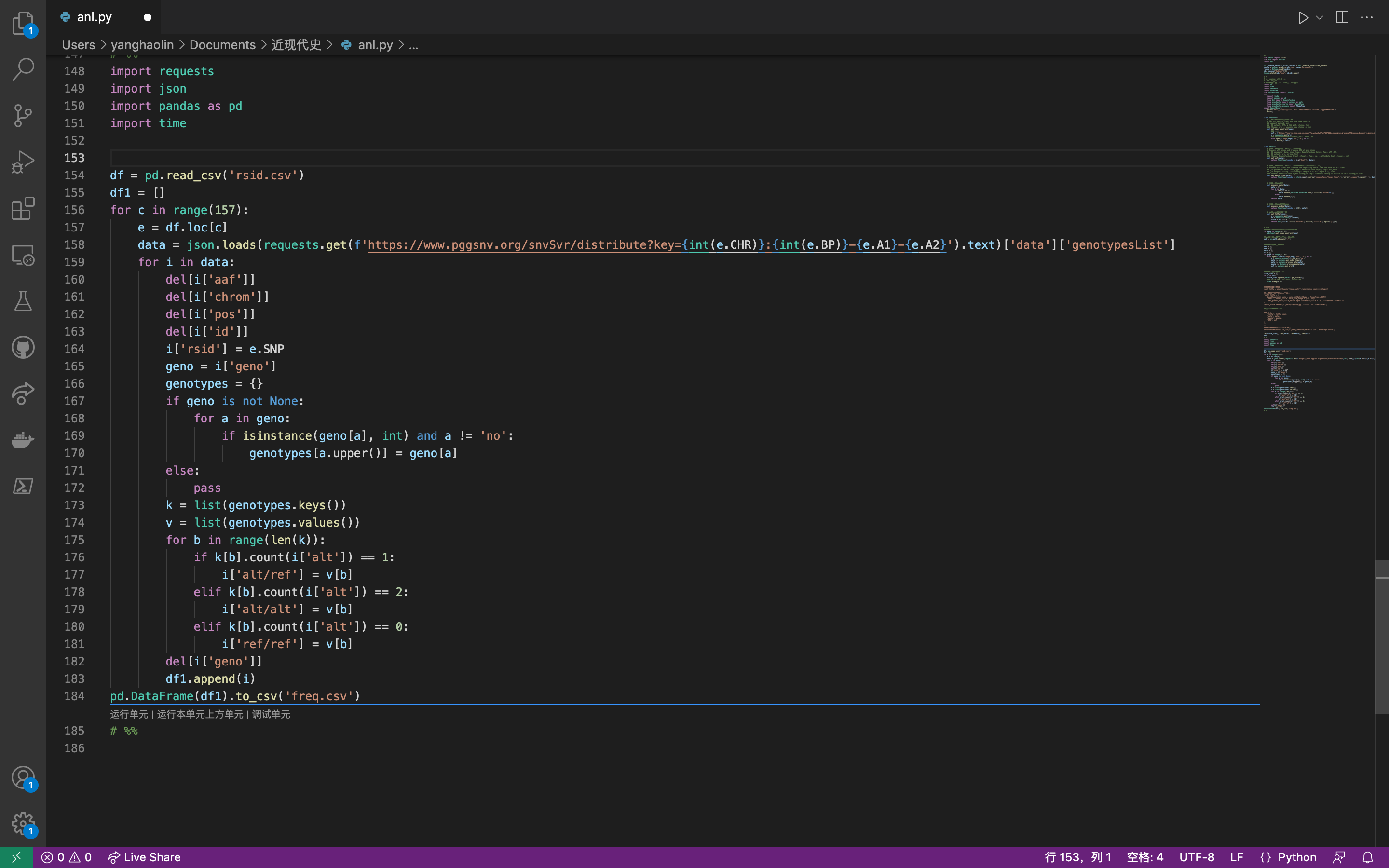
設計出的爬蟲腳本如下:
?
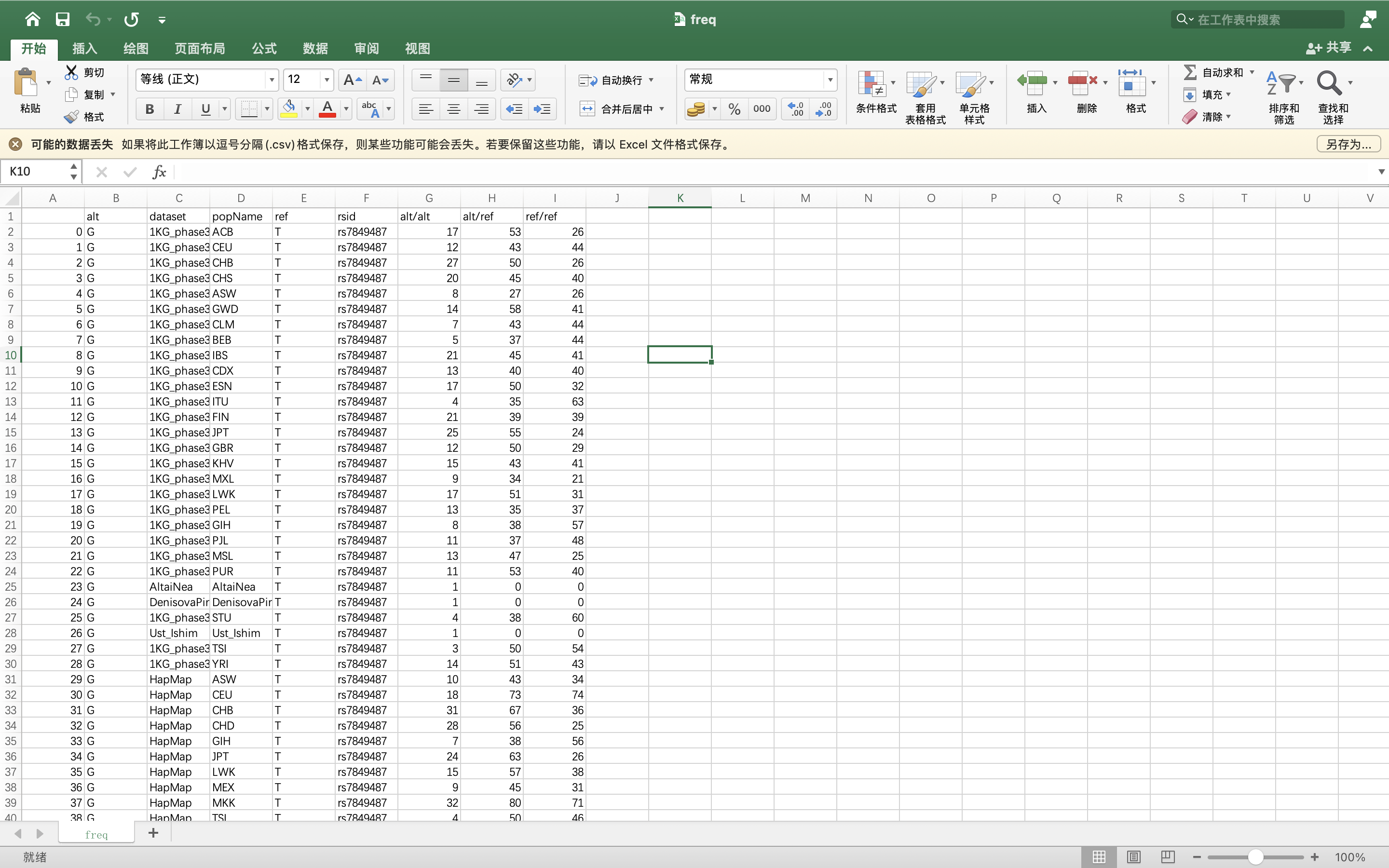
爬取效果如下:
?
reference:
【1】Demange, P.A., Malanchini, M., Mallard, T.T.?et al.?Investigating the genetic architecture of noncognitive skills using GWAS-by-subtraction.?Nat Genet?53,?35–44 (2021). https://doi.org/10.1038/s41588-020-00754-2
【2】Chao Zhang#, Yang Gao#, Zhilin Ning#, Yan Lu#, Xiaoxi Zhang, Jiaojiao Liu, Bo Xie, Zhe Xue, Xiaoji Wang, Kai Yuan, Xueling Ge, Yuwen Pan, Chang Liu, Lei Tian, Yuchen Wang, Dongsheng Lu, Boon-Peng Hoh, Shuhua Xu*. PGG.SNV: understanding the evolutionary and medical implications of human single nucleotide variations in diverse populations. Genome Biology (2019), 20:215.?https://doi.org/10.1186/s13059-019-1838-5
?
代碼:
https://github.com/yhlhhhhh/HGG.SNV_Reptile
?
網站鏈接:https://www.pggsnv.org/
?
但是當我們要查詢的數據量十分龐大時會比較費時間,所以我們寫一段python腳本來批量獲取數據。
首先使用chrome對該網站的基本情況進行分析。使用Chrome的開發者工具對傳輸數據進行監控,并將傳輸數據篩選為fetch和XHR模式,發現該網站的前后端是通過json傳輸的,屬于前后端分離的情況,所以我們可以直接向網站拉去請求獲取json。
?
我們將其中的json打開,發現里面確實含有位點相關信息,所以雙擊該json文件獲取url。
?
url:https://www.pggsnv.org/snvSvr/distribute?key=1:231557623-G-C
?
接著我們對url進行解析。通過對比已知位點數據可知該url中的key應該是以這樣的格式組成的:染色體號:堿基對位置-ref-alt
我們以《Nature-Genetics》期刊上的《Investigating the genetic architecture of noncognitive skills using GWAS-by-subtraction》【1】這篇文章的數據為例
設計出的爬蟲腳本如下:
?
爬取效果如下:
?
reference:
【1】Demange, P.A., Malanchini, M., Mallard, T.T.?et al.?Investigating the genetic architecture of noncognitive skills using GWAS-by-subtraction.?Nat Genet?53,?35–44 (2021). https://doi.org/10.1038/s41588-020-00754-2
【2】Chao Zhang#, Yang Gao#, Zhilin Ning#, Yan Lu#, Xiaoxi Zhang, Jiaojiao Liu, Bo Xie, Zhe Xue, Xiaoji Wang, Kai Yuan, Xueling Ge, Yuwen Pan, Chang Liu, Lei Tian, Yuchen Wang, Dongsheng Lu, Boon-Peng Hoh, Shuhua Xu*. PGG.SNV: understanding the evolutionary and medical implications of human single nucleotide variations in diverse populations. Genome Biology (2019), 20:215.?https://doi.org/10.1186/s13059-019-1838-5
?
代碼:
https://github.com/yhlhhhhh/HGG.SNV_Reptile


2 個回復
贊同來自:
贊同來自:
要回復問題請先登錄或注冊