詳解AlphaFold生成的PDB文件以及3D打印需要的STL文件
繼上篇帖子
http://www.sofreetech.com/question/25230?
里寫的獎預測的模型用3D打印機打印出來,而這其中就涉及到格式轉換的問題,但據我所知貌似沒有專門的轉換工具,所以需要用編程實現。而用編程實現的兩個基礎就是要知道待轉換格式的數據框架和轉換格式的數據框架分別是什么,之后再想解決方案。
--------------------
PDB文件部分:
首先我百度了一下,發現網上給的資料并不多,有些給出的則是比較籠統的解釋,并沒有針對某個實例。這是找到的資料:
https://blog.sciencenet.cn/blog-548663-895916.html
于是我就想通過某種方式打開這個文件并輸出到txt上。受以前繪制發育樹貼:
http://www.sofreetech.com/question/24154
的啟發,我發現雖然biopython要求的是Newick格式,但是把形如Newick的數據框架放在txt中同樣能輸出結果,我就想到既然能夠直接用txt讀取,那為什么不能試試以txt的形式打開呢,于是我打開VScode,寫了這一段Python腳本:
輸出結果發現竟然成功了:
于是我又寫了下面一段腳本將結果輸出到txt中以方便觀察:
輸出結果:
?
首先根據我目前已有的生物和化學知識可以直接看出:
第一列代表的是微粒類型,ATOM代表原子,TER代表殘基
第二列很明顯是微粒編號
第三列嘛。。。不知道,先跳過去,一會再看
第四列,氨基酸名稱,并且是三字母縮寫
第五列,不知道,先跳過
第六列,一看和氨基酸名稱的分布相同,并且有向下標序號的感覺,所以猜是氨基酸序列編號
之后看到后面有5列數,其中第3列仿佛和第4列有些分開,所以我猜7,8,9列是一組數據,10,11列是另一種或兩種數據。我又看了看7,8,9列都有負有正,況且pdb文件中肯定要有表示位置的數據,所以我猜測7,8,9列分別代表著x,y,z坐標。至于10,11列是什么不知道,也先跳過
最后一列,我一看都是字母,而且大多是CHON,所以是元素符號
之后我就查閱了資料,說第三列是原子名稱,第五列是肽鏈名,第11列是占有率,第12列是溫度因子
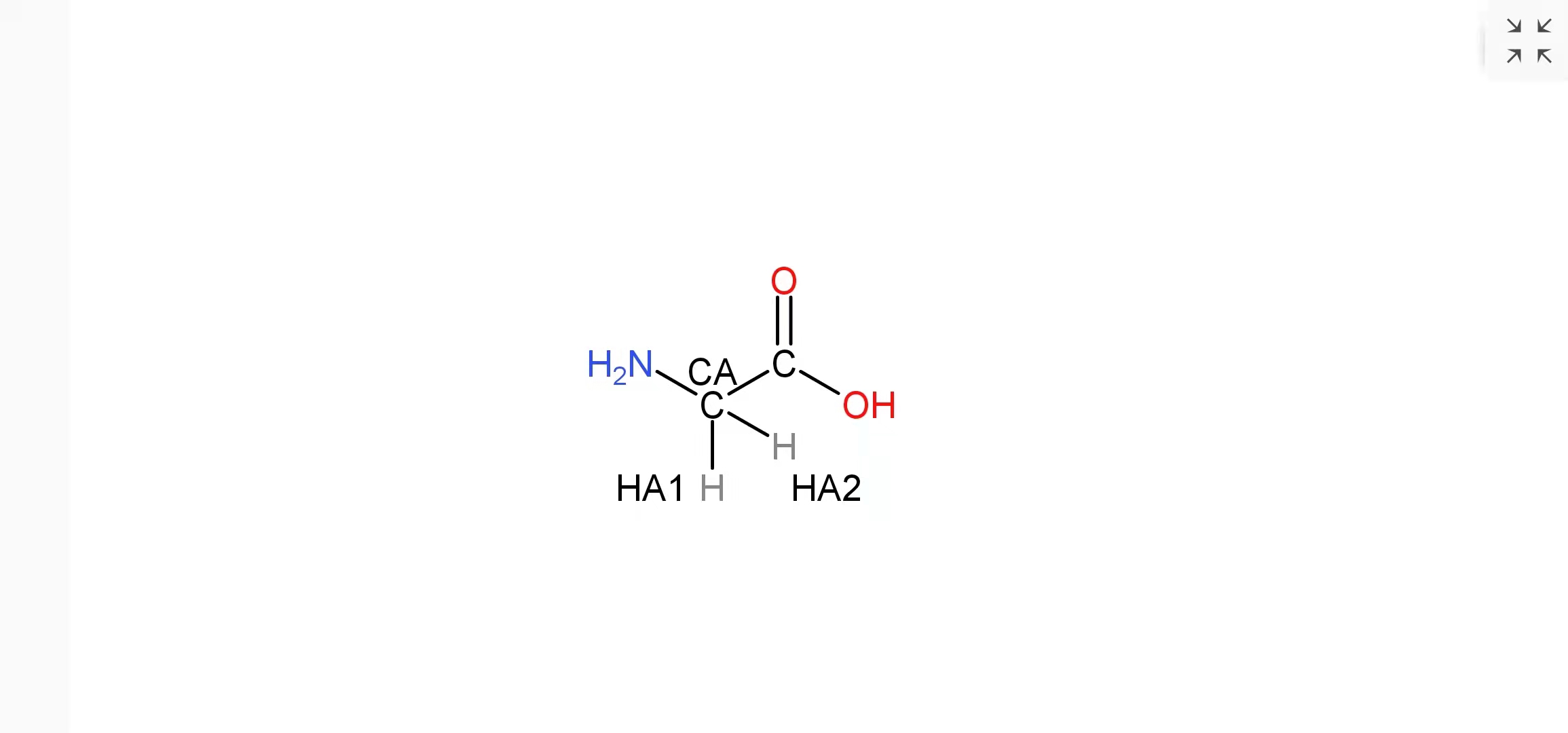
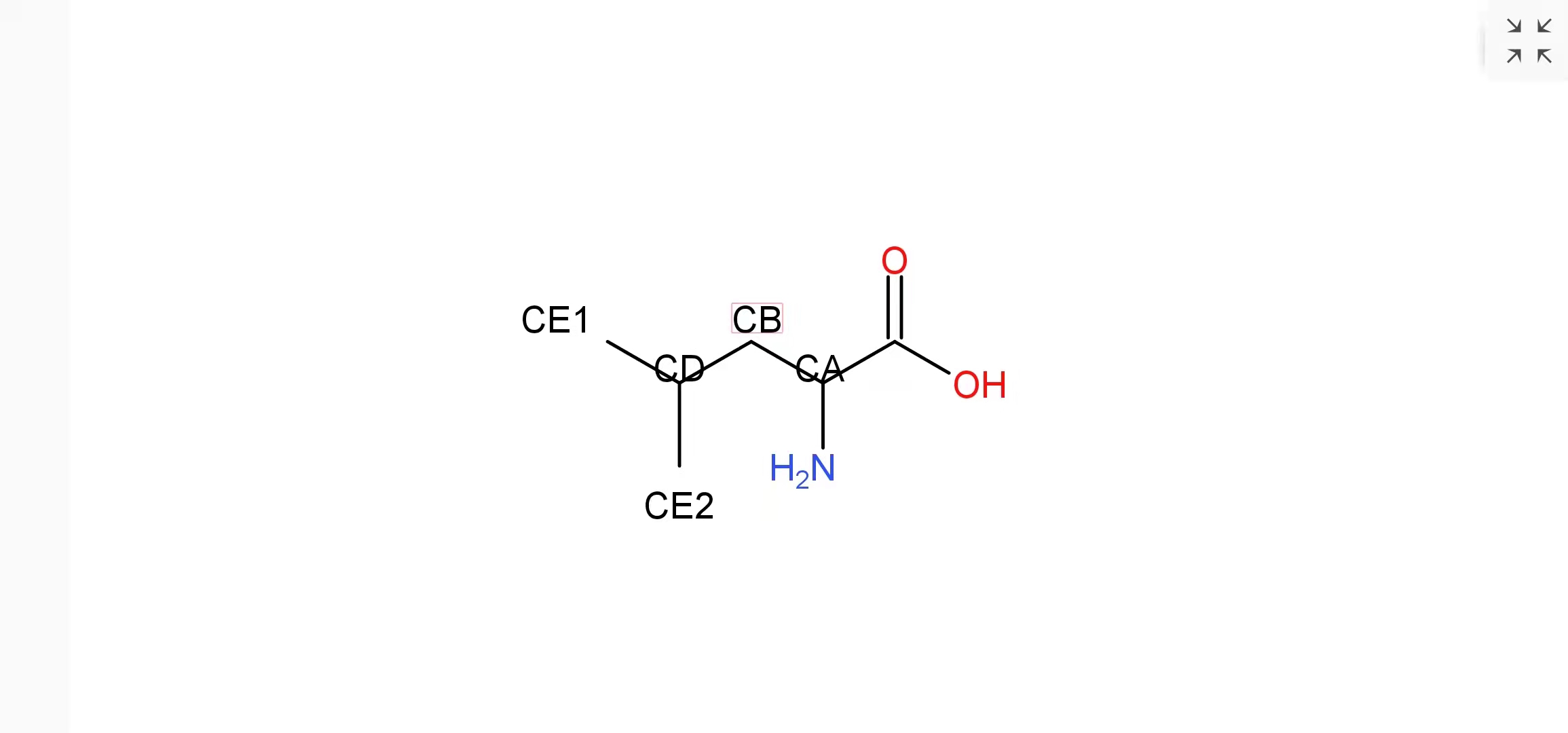
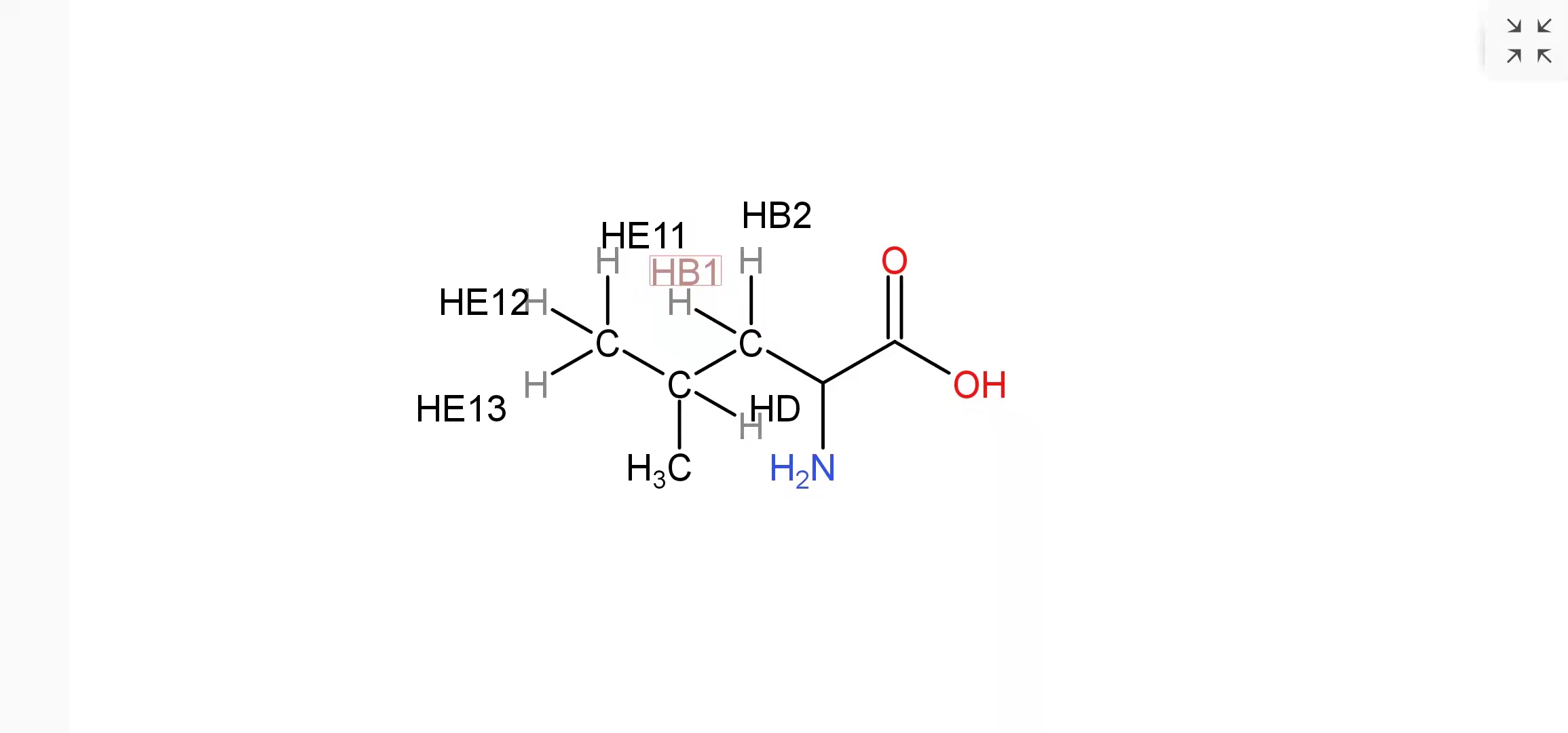
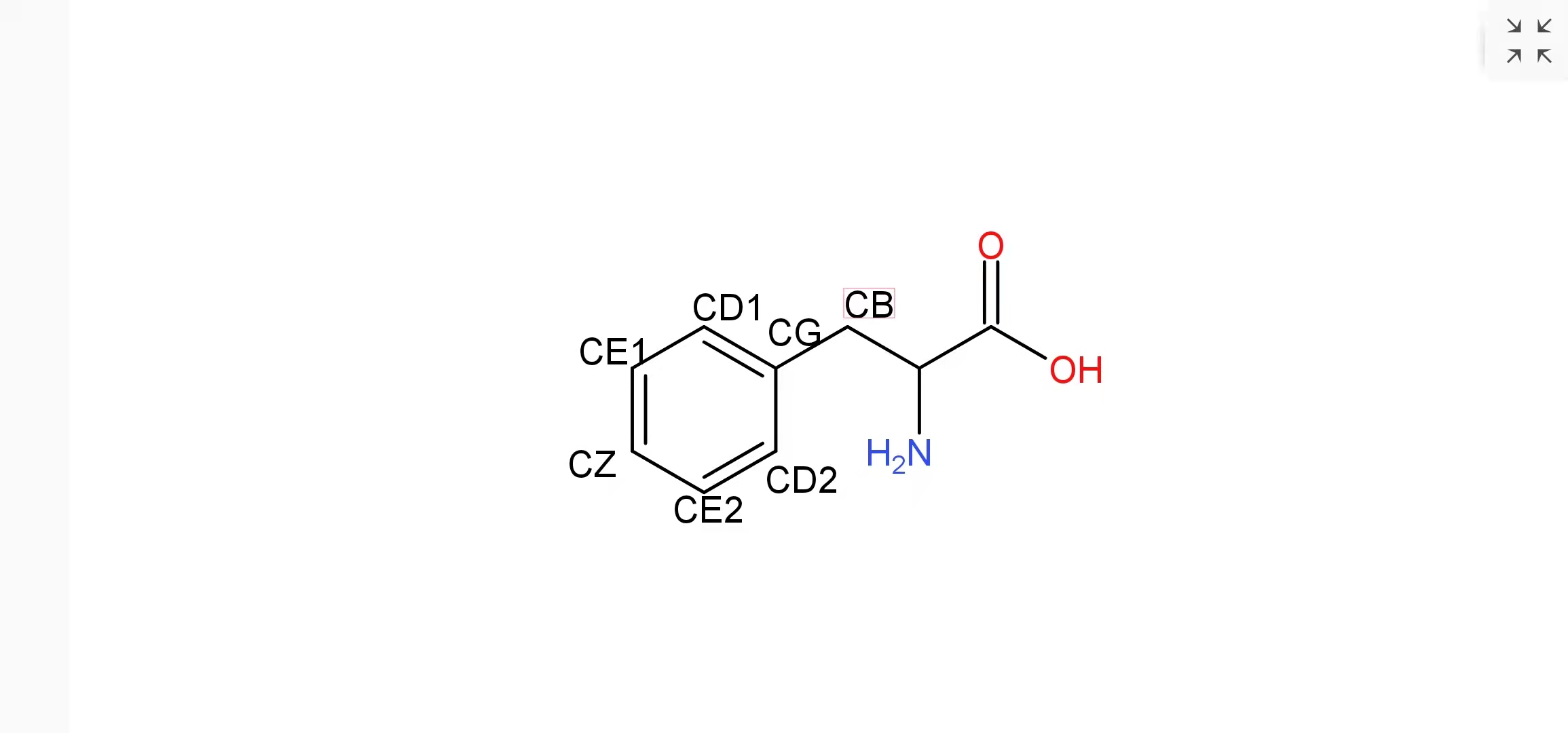
通過我的感覺以及不斷嘗試,我發現第3列是帶著位置信息的原子名稱,規則如下:
第三列表示原子在中心碳或R基的位置
肽鍵上的原子直接寫元素名稱即可
字符第一位為元素名稱
第二位為從中心碳開始到遠中心碳端的距離(從B~Y,不包括C,G)
當R基上存在苯環時,連接苯環的近中心碳端字符第二位為G;該位置的對位碳位置字符第二位則為Z;兩臨位碳字符第二位按照順序向下標,并在第三位分別加上1和2以區分兩個臨位碳;兩對位碳的標注方法與臨位碳相同
若出現4位的情況,則說明R基上有支鏈,此時第3位代表的是主鏈或支鏈,第4位則代表的才是各個氫原子。
?
emmmm這么說可能比較抽象,我舉幾個例子吧:
?
甘氨酸:
?
亮氨酸:
?
苯丙氨酸:
?
-----------------
STL文件部分:
根據我當時在清華做項目的那段時間教授跟我說的,STL有明碼形式和二進制形式,它會將表面用三角來代替以方便打印。但是這執導的信息太少了,于是,我一去問了一下度娘:
二進制STL文件用固定的字節數來給出三角面片的幾何信息。
文件起始的80個字節是文件頭,用于存貯文件名;
緊接著用 4 個字節的整數來描述模型的三角面片個數,
后面逐個給出每個三角面片的幾何信息。每個三角面片占用固定的50個字節,依次是:
3個4字節浮點數(角面片的法矢量)
3個4字節浮點數(1個頂點的坐標)
3個4字節浮點數(2個頂點的坐標)
3個4字節浮點數(3個頂點的坐標)個
三角面片的最后2個字節用來描述三角面片的屬性信息。
一個完整二進制STL文件的大小為三角形面片數乘以 50再加上84個字節。
?
UINT8//Header//文件頭
UINT32//Numberoftriangles//三角面片數量
//foreachtriangle(每個三角面片中)
REAL32[3]//Normalvector//法線矢量
REAL32[3]//Vertex1//頂點1坐標
REAL32[3]//Vertex2//頂點2坐標
REAL32[3]//Vertex3//頂點3坐標
UINT16//Attributebytecountend//文件屬性統計
?
嘿嘿,謝謝度娘(doge
http://www.sofreetech.com/question/25230?
里寫的獎預測的模型用3D打印機打印出來,而這其中就涉及到格式轉換的問題,但據我所知貌似沒有專門的轉換工具,所以需要用編程實現。而用編程實現的兩個基礎就是要知道待轉換格式的數據框架和轉換格式的數據框架分別是什么,之后再想解決方案。
--------------------
PDB文件部分:
首先我百度了一下,發現網上給的資料并不多,有些給出的則是比較籠統的解釋,并沒有針對某個實例。這是找到的資料:
https://blog.sciencenet.cn/blog-548663-895916.html
于是我就想通過某種方式打開這個文件并輸出到txt上。受以前繪制發育樹貼:
http://www.sofreetech.com/question/24154
的啟發,我發現雖然biopython要求的是Newick格式,但是把形如Newick的數據框架放在txt中同樣能輸出結果,我就想到既然能夠直接用txt讀取,那為什么不能試試以txt的形式打開呢,于是我打開VScode,寫了這一段Python腳本:
輸出結果發現竟然成功了:
于是我又寫了下面一段腳本將結果輸出到txt中以方便觀察:
輸出結果:
?
首先根據我目前已有的生物和化學知識可以直接看出:
第一列代表的是微粒類型,ATOM代表原子,TER代表殘基
第二列很明顯是微粒編號
第三列嘛。。。不知道,先跳過去,一會再看
第四列,氨基酸名稱,并且是三字母縮寫
第五列,不知道,先跳過
第六列,一看和氨基酸名稱的分布相同,并且有向下標序號的感覺,所以猜是氨基酸序列編號
之后看到后面有5列數,其中第3列仿佛和第4列有些分開,所以我猜7,8,9列是一組數據,10,11列是另一種或兩種數據。我又看了看7,8,9列都有負有正,況且pdb文件中肯定要有表示位置的數據,所以我猜測7,8,9列分別代表著x,y,z坐標。至于10,11列是什么不知道,也先跳過
最后一列,我一看都是字母,而且大多是CHON,所以是元素符號
之后我就查閱了資料,說第三列是原子名稱,第五列是肽鏈名,第11列是占有率,第12列是溫度因子
通過我的感覺以及不斷嘗試,我發現第3列是帶著位置信息的原子名稱,規則如下:
第三列表示原子在中心碳或R基的位置
肽鍵上的原子直接寫元素名稱即可
字符第一位為元素名稱
第二位為從中心碳開始到遠中心碳端的距離(從B~Y,不包括C,G)
當R基上存在苯環時,連接苯環的近中心碳端字符第二位為G;該位置的對位碳位置字符第二位則為Z;兩臨位碳字符第二位按照順序向下標,并在第三位分別加上1和2以區分兩個臨位碳;兩對位碳的標注方法與臨位碳相同
若出現4位的情況,則說明R基上有支鏈,此時第3位代表的是主鏈或支鏈,第4位則代表的才是各個氫原子。
?
emmmm這么說可能比較抽象,我舉幾個例子吧:
?
甘氨酸:
?
亮氨酸:
?
苯丙氨酸:
?
-----------------
STL文件部分:
根據我當時在清華做項目的那段時間教授跟我說的,STL有明碼形式和二進制形式,它會將表面用三角來代替以方便打印。但是這執導的信息太少了,于是,我一去問了一下度娘:
二進制STL文件用固定的字節數來給出三角面片的幾何信息。
文件起始的80個字節是文件頭,用于存貯文件名;
緊接著用 4 個字節的整數來描述模型的三角面片個數,
后面逐個給出每個三角面片的幾何信息。每個三角面片占用固定的50個字節,依次是:
3個4字節浮點數(角面片的法矢量)
3個4字節浮點數(1個頂點的坐標)
3個4字節浮點數(2個頂點的坐標)
3個4字節浮點數(3個頂點的坐標)個
三角面片的最后2個字節用來描述三角面片的屬性信息。
一個完整二進制STL文件的大小為三角形面片數乘以 50再加上84個字節。
?
UINT8//Header//文件頭
UINT32//Numberoftriangles//三角面片數量
//foreachtriangle(每個三角面片中)
REAL32[3]//Normalvector//法線矢量
REAL32[3]//Vertex1//頂點1坐標
REAL32[3]//Vertex2//頂點2坐標
REAL32[3]//Vertex3//頂點3坐標
UINT16//Attributebytecountend//文件屬性統計
?
嘿嘿,謝謝度娘(doge







0 個回復
要回復問題請先登錄或注冊